Caractéristiques, structures et fonctions des protéines SSB
- 2655
- 812
- Mlle Ambre Dumont
Le Protéines SSB o Bands simples de liaison à l'ADN (de l'anglais "saine-sADN trand bInding Proteins «), Ce sont des protéines responsables de la stabilisation, de la protection et du maintien temporairement.
Les informations génétiques d'un organisme sont protégées et codées dans l'ADN à double bande. Pour que cela puisse être traduit et reproduit, il est nécessaire qu'il se déroule et le chômage et c'est dans ce processus où les protéines SSB participent.
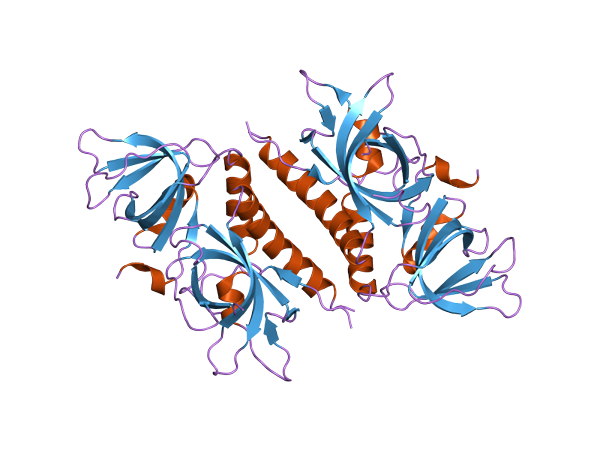
 32 KDA Fragment (RPA32) d'une sous-unité de la protéine de réplication (Source: Jawahar Swaminathan et le personnel MSD à l'Institut européen de bioinformatique [Domaine public] via Wikimedia Commons via Wikimedia)
32 KDA Fragment (RPA32) d'une sous-unité de la protéine de réplication (Source: Jawahar Swaminathan et le personnel MSD à l'Institut européen de bioinformatique [Domaine public] via Wikimedia Commons via Wikimedia) Ces protéines sont cuites en coopération avec d'autres monomères différents qui participent à la stabilisation de leur ADN et se trouvent dans les procaryotes et les eucaryotes.
Protéines SSB de Escherichia coli (ECSSB), ont été les premières protéines décrites de ce type. Ceux-ci ont été caractérisés par fonctionnellement et structurellement et depuis leur découverte, ils ont été utilisés comme modèle d'étude pour ce type de protéine.
Les organismes eucaryotes ont des protéines similaires aux protéines SSB des bactéries, mais chez les eucaryotes, celles-ci sont appelées protéines RPA ou protéines de réplication A (de l'anglais Réplication des protéines A) qu'ils sont fonctionnellement similaires au SSB.
Depuis sa découverte, des modèles biochimiques fonctionnels informatiques ont été utilisés pour l'étude des interactions entre les protéines SSB et l'ADN de chaîne simple afin d'élucider sa fonction dans les processus essentiels du génome de différents organismes.
[TOC]
Caractéristiques
Ce type de protéine se trouve dans tous les royaumes de la vie et bien qu'ils partagent les mêmes propriétés fonctionnelles, elles sont structurellement différentes, en particulier en termes de changements conformationnels, qui semblent être spécifiques pour chaque type de protéine SSB.
Peut vous servir: Système ABO: incompatibilité, héritage et preuveIl a été observé que toutes ces protéines partagent un domaine conservé qui est impliqué dans l'union à l'ADN de la bande simple et qui est connu sous le nom de domaine de l'Union oligonucléotide / oligosaccharides (il se trouve dans la bibliographie sous forme de domaine Ob).
Protéines SSB des bactéries thermophiles telles que Thermus aquaticus Ils ont des caractéristiques remarquables, car ils ont deux domaines OB dans chaque sous-unité, tandis que la plupart des bactéries n'ont qu'un seul dans chaque sous-unité.
La plupart des protéines SSB ne se lient pas spécifiques à l'ADN de la bande simple. Cependant, l'union de chaque SSB dépend de sa structure, de son degré de coopération, du niveau d'oligomérisation et de diverses conditions environnementales.
La concentration d'ions magnésium divalent, la concentration de sels, le pH, la température, la présence de polyamines, de spermidine et de sperme, sont quelques-unes des conditions environnementales étudiées In vitro que le plus affecte l'activité des protéines SSB.
Structure
Les bactéries ont des protéines SSB homo-tétramériques, et chaque sous-unité a une seule maîtrise de l'union OB. Au contraire, les protéines SSB virales, en particulier celles de nombreux bactériophages, sont généralement mono- ou dimétriques.
À son extrémité N-terminale, les protéines SSB ont le domaine de la jonction d'ADN, tandis que leur extrémité C-terminale est composée de neuf acides aminés préservés en charge des interactions protéine-protéine.
Trois résidus de triptophane dans les positions 40, 54 et 88 sont les résidus responsables de l'interaction avec l'ADN dans les domaines syndicaux. Ceux-ci méritent non seulement la stabilisation de l'interaction ADN-protéine, mais aussi le recrutement des autres sous-unités protéiques.
Protéine SSB de ET. coli Il a été modélisé dans des études de calcul et il a été déterminé qu'il a une structure tétramérique de 74 kDa et qu'elle rejoint l'ADN de la bande simple grâce à l'interaction coopérative de différentes sous-unités de type SSB.
Peut vous servir: rouge de phénol: caractéristiques, préparation, applicationsLes archaeas ont également des protéines SSB. Ce sont monomères et n'ont qu'une seule maîtrise de l'ADN ou du domaine OB.
Dans les eucaryotes, les protéines RPA sont structurellement parlant, plus complexes: elles sont constituées d'un hétérotromer (de trois sous-unités différentes) connues sous le nom de RPA70, RPA32 et RPA14.
Ils ont au moins six domaines d'oligonucléotides / oligosaccharides, bien qu'aujourd'hui seulement quatre de ces sites soient connues précisément: trois dans la sous-unité RPA70, et une salle qui réside dans la sous-unité RPA32.
Les fonctions
Les protéines SSB ont des fonctions clés dans l'entretien, l'emballage et l'organisation du génome en protégeant et en stabilisant les brins d'ADN de chaîne simple au moment où ils sont exposés par l'action d'autres enzymes.
Il est important de garder à l'esprit que ces protéines ne sont pas les protéines responsables du déroulement et de l'ouverture des brins d'ADN. Sa fonction n'est limitée que pour stabiliser l'ADN lorsqu'il est à l'état de l'ADN de la bande simple.
Ces protéines SSB agissent en coopération, car l'union de l'un d'eux facilite l'union d'autres protéines (SSB ou non). Dans les processus métaboliques de l'ADN, ces protéines sont considérées comme une sorte de protéines pionnières ou primaires.
L'union de ces protéines à l'ADN a comme fonction principale, en plus de stabiliser les bandes d'ADN de chaîne simple, la protection de ces molécules à la dégradation par les endonucléases de type V.
Les protéines de type SSB participent activement à pratiquement tous les processus d'ADN des organismes vivants. De telles protéines avancent à mesure que la fourche de réplication avance et maintient les deux chaînes d'ADN parentales séparées.
Peut vous servir: lipase pancréatique: structure, fonctions, valeurs normalesExemples
Dans les bactéries, les protéines SSB stimulent et stabilisent les fonctions des protéines. Cette protéine est responsable de la réparation de l'ADN (réaction SOS) et du processus de recombinaison entre des molécules de bande complémentaire simples.
Les mutants de ET. coli Génétiquement manipulé pour obtenir des protéines SSB défectueux, sont rapidement inhibés et ne remplissent pas efficacement leurs fonctions dans la réplication, la réparation et la recombinaison de l'ADN.
Les protéines de type RPA contrôlent la progression du cycle cellulaire dans les cellules eucaryotes. Plus précisément, on pense que la concentration cellulaire de RPA4 pourrait avoir une influence indirecte sur le col de la réplication de l'ADN, c'est-à-dire que dans des concentrations élevées de RPA4, ce processus est inhibé.
Il a été suggéré que l'expression de RPA4 peut empêcher la prolifération cellulaire en inhibant la réplication et en jouant un rôle dans le maintien et le marquage de la viabilité des cellules saines dans les organismes animaux.
Les références
- Anthony, E., & Lohman, t. M. (2019, février). Dynamique de e. Complexes protéine-ADN-ADN-ADN de liaison à l'ADN unique de coli (SSB). Dans Séminaires en biologie cellulaire et développemental (Vol. 86, pp. 102-111). Presse universitaire.
- Beernink, H. T., & Morrical, S. W. (1999). RMPS: Protéines de médiateur de recombinaison / réplication. Tendances des sciences biochimiques, 24(10), 385-389.
- Bianco, P. R. (2017). L'histoire de SSB. Progrès en biophysique et biologie moléculaire, 127, 111-118.
- Byrne, b. M., & Oakley, G. g. (2018, novembre). Protéine A réplication, le laxatif qui maintient l'ADN régulier: l'importance de la phosphorylation de la RPA dans le maintien de la stabilité du génome. Dans Séminaires en biologie cellulaire et développemental. Presse universitaire
- Krebs, J. ET., Goldstein, E. S., & Kilpatrick, s. T. (2017). Gènes de Lewin XII. Jones & Bartlett Learning.
- LeCointe, F., Serena, C., Velten, m., Coûts, un., McGovern, S., Meile, J. C.,… & Pollard, P. (2007). Anticiper l'arrêt de la fourche de réplication chromosomique: SSB cible la réparation des hélicases d'ADN aux fourches actives. Le journal EMBO, 26(19), 4239-4251.
- « Caractéristiques d'amplitude des vagues, formules et exercices
- Caractéristiques, styles et stratégies d'apprentissage actifs »

