Fonctions, types et exemples enzymes de restriction
- 4607
- 663
- Raphaël Meyer
Le les enzymes de restriction Ce sont des endonucléases utilisées par certaines arches et bactéries pour inhiber ou "restreindre" la propagation des virus à l'intérieur. Ils sont particulièrement communs dans les bactéries et font partie de leur système de défense ADN étranger connu sous le nom de système de restriction / modification.
Ces enzymes catalysent la coupe d'ADN à double bande dans des endroits spécifiques, reproductible et sans utiliser d'énergie supplémentaire. La plupart nécessitent la présence de cofacteurs tels que le magnésium ou d'autres cations divalents, bien que certains nécessitent également de l'ATP ou du S-adénosyl metionine.
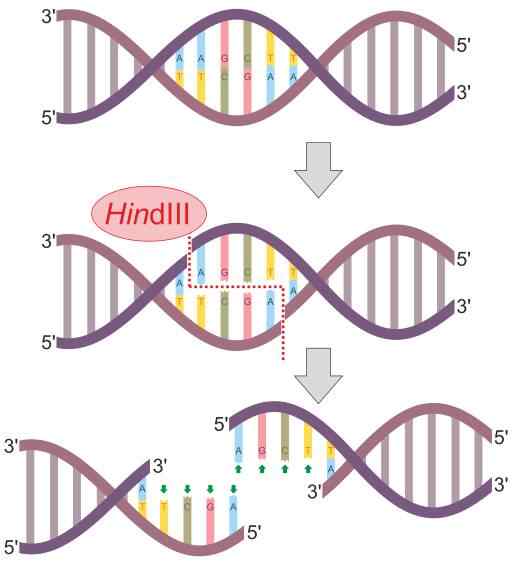
 Schéma de réaction de l'enzyme de restriction de l'hindii (Source: Helixitta [CC BY-SA 4.0 (https: // CreativeCommons.Org / licences / by-sa / 4.0)] via Wikimedia Commons)
Schéma de réaction de l'enzyme de restriction de l'hindii (Source: Helixitta [CC BY-SA 4.0 (https: // CreativeCommons.Org / licences / by-sa / 4.0)] via Wikimedia Commons) Les endonucléases de restriction ont été découvertes en 1978 par Daniel Nathans, Arber Werner et Hamilton Smith, qui a reçu le prix Nobel de médecine pour leur découverte. Son nom dérive généralement de l'organisme où ils sont observés pour la première fois.
Ces enzymes sont largement utilisées dans le développement de méthodes de clonage ADN et d'autres stratégies de biologie moléculaire et de génie génétique. Ses caractéristiques de reconnaissance de séquences spécifiques et de capacité de coupe des séquences proches des sites de reconnaissance en font des outils puissants en expérimentation génétique.
Les fragments générés par les enzymes de restriction qui ont agi sur une molécule d'ADN particulière peuvent être utilisés pour recréer une «carte» de la molécule d'origine en utilisant des informations sur les sites où l'enzyme a coupé l'ADN.
Certaines enzymes de restriction peuvent avoir le même site de reconnaissance dans l'ADN, mais ils ne le coupent pas nécessairement de la même manière. Ainsi, il existe des enzymes qui effectuent des coupes en laissant des romos et des enzymes qui coupent les extrêmes cohésifs, qui ont des applications différentes en biologie moléculaire.
Il existe actuellement des centaines d'enzymes de restriction différentes disponibles dans le commerce, offertes par différentes maisons commerciales; Ces enzymes fonctionnent comme des ciseaux moléculaires «sur mesure» à différentes fins.
[TOC]
Les fonctions
Les enzymes de restriction remplissent la fonction inverse des polymérases, car elles sont hydrolysées ou brisent la liaison ester dans la liaison phosphodiéster entre les nucléotides adjacents dans une chaîne nucléotidique.
En biologie moléculaire et en génie génétique, ils sont des outils largement utilisés pour la construction de vecteurs d'expression et de clonage, ainsi que pour l'identification de séquences spécifiques. Ils sont également utiles pour la construction de génomes recombinants et ont un grand potentiel biotechnologique.
Les progrès récents de la thérapie génique font de l'utilisation actuelle des enzymes de restriction pour l'introduction de gènes déterminés dans des vecteurs qui sont des véhicules pour le transport de ces gènes vers les cellules vivantes, et qu'ils ont probablement la capacité de s'insérer dans le génome cellulaire pour effectuer des changements permanents.
Mécanisme d'action
Les enzymes de restriction peuvent catalyser la coupe d'ADN à double bande, bien que certains soient capables de reconnaître les séquences d'ADN de bande simples et même l'ARN. La coupe se produit après la reconnaissance des séquences.
Peut vous servir: Cycle de glioxylate: caractéristiques, réactions, régulation, fonctionsLe mécanisme d'action consiste en l'hydrolyse du lien phosphodiéster entre un groupe phosphate et un désoxyribose dans le squelette de chaque ADN d'ADN. De nombreuses enzymes sont capables de couper au même endroit qu'elles reconnaissent, tandis que d'autres coupent entre 5 et 9 paires de bases avant ou après la même.
Normalement, ces enzymes se coupent à l'extrémité 5 'du groupe phosphate, donnant naissance à des fragments d'ADN avec une extrémité phosphoryle de 5' et une extrémité hydroxyle 3 'terminal.
Étant donné que les protéines ne sont pas en contact direct avec le site de reconnaissance de l'ADN, celles-ci doivent être transloquées successives jusqu'à ce que le site spécifique soit obtenu, peut-être par des mécanismes de «glissement» sur le brin d'ADN.
Pendant la coupe enzymatique, la liaison phosphodiéster de chacun des brins d'ADN est positionnée dans l'un des sites actifs des enzymes de restriction. Lorsque l'enzyme quitte le site de reconnaissance et de coupe, il le fait par le biais d'associations transitoires non spécifiques.
Gars
Actuellement, cinq types d'enzymes de restriction sont connus. Ensuite, une brève description de chacun:
Enzymes de restriction de type I
Ces enzymes sont de grandes protéines pentamériques avec trois sous-unités, une de restriction, une méthylation et une autre pour la reconnaissance des séquences d'ADN. Ces endonucléases sont des protéines multifonctionnelles capables de catalyser les réactions de restriction et de modification, ont l'activité de l'ATPASA et également de l'ADN topoisomera.
Les enzymes de ce type ont été les premières endonucléases à découvrir, elles ont été purifiées pour la première fois dans les années 1960 et depuis lors, elles ont été étudiées avec une grande profondeur.
Les enzymes de type I ne sont pas largement utilisées comme outil biotechnologique, car le site de coupe peut être à une distance variable jusqu'à 1.000 paires de bases concernant le site de reconnaissance, ce qui les rend peu fiables en termes de reproductibilité expérimentale.
Enzymes de restriction de type II
Ce sont des enzymes composées d'homodimères ou de tétramères qui coupent l'ADN en sites définis entre 4 et 8 pb de longueur. Ces sites de coupe sont généralement palindromiques, c'est-à-dire qu'ils reconnaissent les séquences qui sont lues de la même manière dans les deux directions.
De nombreuses enzymes de restriction de type II dans les bactéries ont coupé l'ADN lorsqu'ils reconnaissent leur caractère étranger, car il n'a pas les modifications typiques que le propre ADN devrait avoir.
Ce sont des enzymes de restriction plus simples car elles ne nécessitent aucun cofacteur autre que le magnésium (mg +) pour reconnaître et couper les séquences d'ADN.
La précision des enzymes de restriction de type II dans la reconnaissance et la coupe de séquences simples dans l'ADN dans des positions précises en fait l'une des plus utilisées et indispensables dans la plupart des branches de la biologie moléculaire.
Peut vous servir: mutualisme: caractéristiques, types, exemplesDans le groupe des enzymes de restriction de type II, il y a plusieurs sous-classes classées en fonction de certaines propriétés qui sont uniques pour chacun. La classification de ces enzymes est effectuée en ajoutant des lettres de l'alphabet, de A à Z suivant le nom de l'enzyme.
Certaines des sous-classes les plus connues pour leur utilité sont:
Sous-classe IIA
Ce sont des sous-unité différentes dímeros. Ils reconnaissent les séquences asymétriques et sont utilisés comme précurseurs idéaux pour la génération d'enzymes de coupe.
Sous-classe IIB
Ils sont composés d'un autre dimères et coupent l'ADN des deux côtés de la séquence de reconnaissance. Ils ont coupé les deux brins d'ADN dans un intervalle de base de base plus tard du site de reconnaissance.
Sous-classe IIC
Les enzymes de ce type sont des polypeptides avec des fonctions de division et de modification des brins d'ADN. Ces enzymes coupent les deux brins asymétriquement.
Sous-classe IIE
Les enzymes de cette sous-classe sont les plus utilisées en génie génétique. Ils ont un site catalytique et nécessitent généralement un effecteur allestro. Ces enzymes doivent interagir avec deux copies de leur séquence de reconnaissance pour effectuer une coupe efficace. Dans cette sous-classe se trouvent les enzymes ECORII et ECORI.
Enzymes de restriction de type III
Les endonucléases de restriction de type III ne sont composées que de deux sous-unités, l'une est responsable de la reconnaissance et de la modification de l'ADN, tandis que l'autre est responsable de la coupe de la séquence.
Ces enzymes nécessitent deux cofacteurs pour leur opération: ATP et magnésium. Les enzymes de restriction de ce type ont deux sites de reconnaissance asymétriques, transloquer l'ADN d'une manière dépendante de l'ATP et la couper entre 20 et 30 pb adjacente au site de reconnaissance.
Enzymes de restriction de type IV
Les enzymes de type IV sont faciles à identifier car elles coupent l'ADN avec des marques de méthylation, elles sont composées de plusieurs sous-unités différentes qui sont responsables de la reconnaissance et de la coupe de la séquence d'ADN. Ces enzymes utilisent des cofacteurs GTP et du magnésium divalent.
Les sites de coupe spécifiques comprennent des chaînes nucléotidiques avec des résidus de cytosine méthylés ou hydroxyméchées dans une ou les deux brins d'acides nucléiques.
Enzymes de restriction de type V
Cette classification groupe des enzymes de type Crisper-Cas, qui identifient et coupent des séquences d'ADN spécifiques d'organismes envahisseurs. Les enzymes CAS utilisent un guide synthétisé par le brin d'ARN pour Crisper pour reconnaître et attaquer les organismes envahisseurs.
Les enzymes classées comme type V sont des polypeptides structurés par des enzymes de type I, II et II. Ils peuvent couper des sections d'ADN de presque tous les organismes et avec une large gamme de longueurs. Sa flexibilité et sa facilité d'emploi font de ces enzymes l'un des outils les plus utilisés en génie génétique actuellement avec des enzymes de type II.
Peut vous servir: propionibacteriumExemples
Des enzymes de restriction ont été utilisées pour la détection des polymorphismes d'ADN, en particulier dans les études de génétique de la population et les études évolutives utilisant l'ADN mitochondrial, afin d'obtenir des informations sur les taux de substitution nucléotidique.
À l'heure actuelle, les vecteurs utilisés pour la transformation des bactéries à diverses fins ont des sites multiclonages où des sites de reconnaissance pour plusieurs enzymes de restriction sont trouvés.
Parmi ces enzymes les plus populaires sont Ecori, II, III, IV et V, obtenus et décrits pour la première fois à partir de ET. coli; Hindiii, de H. grippe et bamhi de B. Amyloliquefaciens.
Les références
- Bikle, t. POUR., & Kruger, D. H. (1993). Biologie de la restriction de l'ADN. Revues microbiologiques, 57(2), 434-450.
- Boyaval, p., Moineau, s., Romero, D. POUR., & Horvath, P. (2007). CRISPR fournit une acquisition contre les virus dans les procaryotes. Science, 315(Mars), 1709-1713.
- Goodsell, D. (2002). La perspective moléculaire: restriction endonucléases. Fondamentaux des cellules souches de la médecine du cancer, vingt, 190-191.
- Halford, S. ET. (2001). Sauter, sauter et boucler par des enzymes de restriction. Transactions de la société biochimique, 29, 363-373.
- Jeltsch, un. (2003). Maintien de l'identité des espèces et contrôlant la spéciation des bactéries: une nouvelle fonction pour les systèmes de restriction / modification? Gène, 317, 13-16.
- Krebs, J., Goldstein, E., & Kilpatrick, s. (2018). Gènes de Lewin XII (12 ed.). Burlington, Massachusetts: Jones & Bartlett Learning.
- Li, et., Pain, s., Zhang, et., Ren, M., Feng, m., Peng, n.,... elle, q. (2015). Exploitation de systèmes CRISPR-CAS de type I et de type III pour l'édition du génome. Recherche des acides nucléiques, 1-12.
- Loenen, W. POUR. M., Dryden, D. T. F., Raleight, E. POUR., & Wilson, G. g. (2013). Enzymes de restriction de type I et leurs proches. Recherche des acides nucléiques, 1-25.
- Nathans, D., & Smith, H. SOIT. (1975). Endonucléases de restriction dans l'analyse et la restructuration des molécules d'ADN. Anneau. Tour. Biochimie., 273-293.
- Nei, m., & Tajima, F. (1981). Polymorphisme d'ADN détectable par restriction endonucléases. La génétique, 145-163.
- Pingoud, un., Fuxreiter, m., Pingoud, V., & Wende, w. (2005). Endonucléases de restriction de la vie cellulaire et moléculaire de type II: structure et mécanisme. CMLS Sciences de la vie cellulaire et moléculaire, 62, 685-707.
- Roberts, R. (2005). Comment les enzymes de restriction sont devenues les chevaux de travail de la biologie moléculaire. PNA, 102(17), 5905-5908.
- Roberts, R. J., & Murray, K. (1976). Endonucléases de restriction. Critiques critiques en biochimie, (Novembre), 123-164.
- Stoddard, b. L. (2005). Structure et fonction des endonucléases à domicile. Revues trimestrielles de la biophysique, 1-47.
- Tock, m. R., & Dryden, D. T. F. (2005). La biologie de la restriction et de la anti-restauration. Opinion actuelle en microbiologie, 8, 466-472. https: // doi.org / 10.1016 / j.mon B.2005.06.003
- Wilson, G. g., & Murray, n. ET. (1991). Systèmes de restriction et de modification. Anneau. Tour. Génétique., 25, 585-627.
- Wu, z., & Mou, k. (2016). Aperçu génomique sur la virulence de Campylobacter Jejuni et la génétique de la population. Infec. Dis. Traduire. Médicament., 2(3), 109-19.
- Yuan, R. (1981). Structure et mécanisme des endonucléases de restriction multifonctionnelles. Anneau. Tour. Biochimie., cinquante, 285-315.
- « Génération de caractéristiques, auteurs et œuvres de 1914
- Propriétés et exemples d'événements mutuellement exclusifs »

