Traduction du processus d'ADN dans les eucaryotas et les procaryotes
- 4071
- 90
- Raphaël Charles
La Traduction d'ADN C'est le processus par lequel les informations contenues dans les messagers produits pendant la transcription.
Du point de vue cellulaire, l'expression d'un gène est une question relativement complexe qui se produit en deux étapes: transcription et traduction.
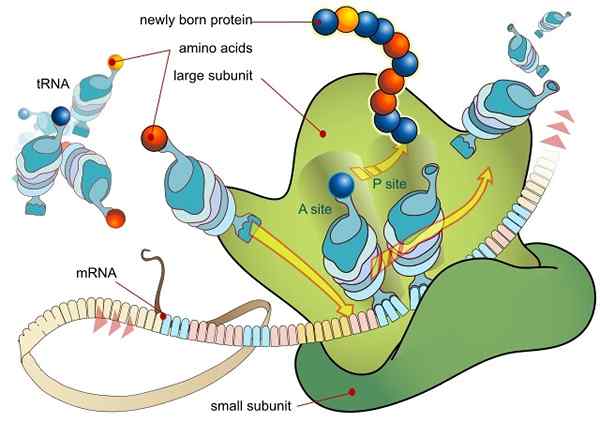
 Traduction d'ARN médiée par un ribosome (source: Ladyofhats / Domaine public, via Wikimedia Commons)
Traduction d'ARN médiée par un ribosome (source: Ladyofhats / Domaine public, via Wikimedia Commons) Tous les gènes qui sont exprimés (que ce soit le codage pour les séquences peptidiques, c'est-à-dire les protéines) le font initialement en transférant les informations contenues dans sa séquence d'ADN à une molécula Mer Mer Messager (RNAM) via un processus appelé transcription.
La transcription est réalisée par des enzymes spéciales appelées ARN polymérases, qui utilisent l'un des brins complémentaires de l'ADN du gène comme moule pour la synthèse d'une molécule «pré-arm», qui est par la suite poursuivie pour former une maturité mature.
Pour les gènes qui codent pour les protéines, les informations contenues dans le RNM mature sont «lues» et traduites en acides aminés selon le code génétique, qui spécifie quel codon ou nucléotide triplet correspond à chaque acide aminé particulier.
La spécification de la séquence d'acides aminés d'une protéine dépend donc de la séquence initiale de bases d'azote dans l'ADN qui correspond au gène puis dans l'ARN qui transporte ces informations du noyau au cytosol (dans les cellules eucaryotes); processus qui est également défini comme la synthèse des protéines guidées par RNM.
Compte tenu du fait qu'il existe 64 combinaisons possibles des 4 bases d'azote qui forment l'ADN et l'ARN et seulement 20 acides aminés, un acide aminé peut être codé par différents triplets (codons), il est donc dit que le code génétique est " dégénéré "(à l'exception de la métitionnine d'acide aminé, qui est codée par un codon AUG unique).
[TOC]
Traduction d'Eucaryota (étapes-procédures)
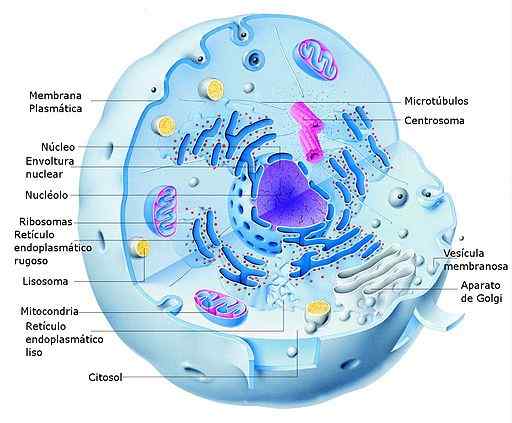
 Diagramme d'une cellule eucaryot animale et de ses parties (Source: Alejandro Porto [CC0] via Wikimedia Commons)
Diagramme d'une cellule eucaryot animale et de ses parties (Source: Alejandro Porto [CC0] via Wikimedia Commons) Dans les cellules eucaryotes, la transcription se déroule dans le noyau et la traduction dans le cytosol, de sorte que les RNM qui se forment au cours du premier processus remplissent également une fonction dans le transport des informations du noyau vers le cytosol, où la machinerie biosynthétique (ribosomes ).
Il est important de mentionner que la compartimentation de la transcription et de la traduction chez les eucaryotes est vraie pour le noyau, mais ce n'est pas la même.
Les cellules eucaryotes ont également des ribosomes cytosoliques attachés aux membranes du réticulum endoplasmique (réticulum endoplasmique rugue.
- Traitement RNM avant leur traduction
Les RNM sont modifiés à leurs extrémités lorsqu'ils sont transcrits:
- Lorsque l'extrémité 5 'du RNM émerge de la surface de l'ARN polymérase II pendant la transcription, ceci est "attaqué" immédiatement par un groupe d'enzymes qui synthétisent un "capot" composé de 7-méthyl guanilado et qui est connecté au terminal nucléotide du RNM à travers une liaison triffosphate de 5 ', 5'.
Peut vous servir: codon- La fin du 3 'du RNM souffre d'un "Clivaje" par une endonucléase, qui génère un groupe hydroxyle gratuit 3' qui rejoint un "Ristra" ou "queue" de déchets d'adénine (de 100 à 250) qui en sont ajoutés un à la même chose temps pour une enzyme Poly (A) polymérase.
Le "Hood 5 '" et la "queue Poly A », ils remplissent les fonctions dans la protection des molécules RNM contre la dégradation et, en outre, ils travaillent dans le transport de transcriptions matures vers le cytosol et dans l'initiation et la terminaison de la traduction, respectivement.
COrte et empalme
Après la transcription, les RNM «primaires» avec leurs deux extrêmes modifiés, toujours présents dans le noyau, passent à travers un processus «coupé et épissage» par lequel les séquences introniques sont généralement éliminées et les exons résultants sont joints (traitement post-régional), avec ce que les transcriptions matures qui abandonnent le noyau et atteignent le cytosol sont obtenues.
La coupe et l'épissage sont réalisés par un complexe riboprotéique appelé le Espliccleosoma (Anglicisme de Épisser), formé par cinq petites ribonucléoprotéines et molécules d'ARN, qui sont capables de "reconnaître" les régions qui doivent être éliminées de la transcription primaire.
Dans de nombreux eucaryotes, il existe un phénomène connu sous le nom de «coupe et articulation alternative», ce qui signifie que différents types de modifications post-consistationnelles peuvent provoquer des protéines différentes ou isoenzymes qui diffèrent les unes des autres à certains aspects de leurs séquences.
- Les ribosomes
Lorsque les transcrits matures quittent le noyau et sont transportés pour la traduction dans le cytosol, ceux-ci sont traités par le complexe de translation appelé ribosome, qui se compose d'un complexe protéique associé aux molécules d'ARN.

Les ribosomes sont composés de deux sous-unités, un "grand" et un autre "petit", qui sont librement dissociés dans le cytosol et se joignent ou s'associent sur la molécule d'ARNm qui se traduit.
L'union entre les ribosomes et l'ARNm dépend de molécules d'ARN spécialisées qui sont associées aux protéines ribosomales (ARN ribosomal ou ARN et transfert ou ARN ARNT), dont chacun exerce des fonctions spécifiques.
Les ARNT sont des "adaptateurs" moléculaires, car à l'une de leurs extrémités, ils peuvent "lire" chaque codon ou triplet dans l'ARN mature (par complémentarité des bases) et à travers l'autre, ils peuvent rejoindre l'acide aminé codé par le codon "Read".
Les molécules RNR, en revanche, sont responsables de l'accélération (catalyser) le processus de liaison de chaque acide aminé dans la chaîne peptidique naissante.
Un RNM mature eucaryote peut être "lu" par de nombreux ribosomes, autant de fois que la cellule l'indique. En d'autres termes, le même RNM peut conduire à de nombreuses copies de la même protéine.
Codon de démarrage et cadre de lecture
Lorsqu'un RNM mature est approché par les sous-unités ribosomales, le complexe riboprotéique "scan" la séquence de ladite molécule jusqu'à ce qu'un codon de démarrage trouve, qui est toujours AUG et implique l'introduction d'un résidu de méthionine.
Il peut vous servir: Monoploïdie: comment cela se produit, les organismes, la fréquence et l'utilitéLe codon AUG définit le cadre de lecture pour chaque gène et, en outre, définit le premier acide aminé de toutes les protéines traduit dans la nature (cet acide aminé est souvent éliminé après la translation)).
Codons de terminaison
Trois autres codons ont été identifiés comme ceux qui induisent la résiliation de la traduction: UAA, UAG et UGA.
Ces mutations qui impliquent un changement de bases d'azote dans le triplet qui code pour un acide aminé et qui entraînent des codons de terminaison sont appelées mutations sans signification, car elles provoquent une détention prématurée du processus de synthèse, qui forme des protéines plus courtes plus courtes.
Régions non traduites
Près de la fin des 5 'des molécules RNM matures, il existe des régions qui ne sont pas traduites (UTR, de l'anglais Région non traduite), également appelées séquences «leader», qui sont situées entre le premier nucléotide et le début de la traduction (août).
Ces régions UTR qui ne sont pas traduites ont des sites spécifiques pour l'union avec des ribosomes et des humains, par exemple, ils ont une longueur approximative de 170 nucléotides, parmi lesquels il existe des régions régulateurs, des sites de liaison aux protéines qui fonctionnent dans la régulation de la traduction, etc.
- Début de la traduction
La traduction, ainsi que la transcription, se compose de 3 phases: l'une des initiations, une autre allongement et enfin une de résiliation.
Initiation
Il se compose de l'assemblage du complexe de translation sur le RNM, qui mérite l'union de trois protéines appelées facteurs d'initiation (si, de l'anglais Facteur d'initiation) If1, if2 et if3 à la petite sous-unité du ribosome.
Le complexe "pré-initiation" formé par les facteurs d'initiation et la petite sous-unité ribosomale sont joints, à leur tour, avec un art qui "charge" un résidu de métification et cet ensemble de molécules se lie au RNAM, près du codon de départ août.
Ces événements conduisent à l'Union RNM avec la grande sous-unité ribosomale, ce qui conduit à la libération de facteurs d'initiation. La grande sous-unité du ribosome a 3 sites d'union pour les molécules ARNT: site A (acide aminé), site P (polypeptide) et site E (sortie).
Le site A rejoint l'anticod de l'Aminoacil-Arnt, qui est complémentaire à celui de l'ARNm qui est traduit; Le site P est l'endroit où l'acide aminé est transféré de l'ARNT au peptide naissant et le site S est où il est en ARNT «vide» avant d'être libéré au cytosol après avoir livré l'acide aminé.
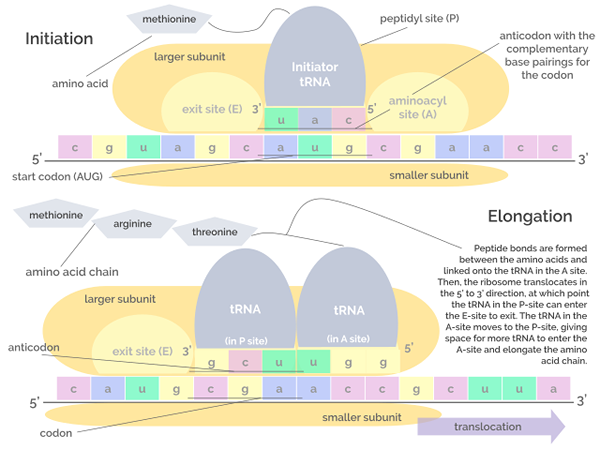 Représentation graphique des phases d'initiation et d'allongement de la traduction (Source: Jordan Nguyen / CC By-S (https: // CreativeCommons.Org / licences / by-sa / 4.0) via Wikimedia Commons)
Représentation graphique des phases d'initiation et d'allongement de la traduction (Source: Jordan Nguyen / CC By-S (https: // CreativeCommons.Org / licences / by-sa / 4.0) via Wikimedia Commons) Élongation
Cette phase se compose du "mouvement" du ribosome le long de la molécule d'ARNm et de la traduction de chaque codon que "la lecture" ce qui implique la croissance ou l'allongement de la chaîne polypeptidique à la naissance.
Ce processus nécessite un facteur connu sous le nom de facteur d'allongement du GTP sous la forme de GTP, qui est celui qui entraîne la translocation des facteurs d'allongement le long de la molécule RNM pendant qu'il traduit.
Peut vous servir: fragments okazakiL'activité peptidil transférase des ARN ribosomales permet la formation de liaisons peptidiques entre les acides aminés successifs qui sont ajoutés à la chaîne.
Résiliation
La traduction se termine lorsque le ribosome rencontre l'un des codons de terminaison, car les ARN ne reconnaissent pas ces codons (ils ne codent pas les acides aminés). Les protéines appelées facteurs de libération sont également jointes, ce qui facilite le détachement du ribosoma MR.
Traduction du procréation (passes-processus)
Dans les procaryotes, comme dans les cellules eucaryotes, les ribosomes en charge de la synthèse des protéines se trouvent dans le cytosol (ce qui est également vrai pour les machines transcriptionnelles), un fait qui permet une augmentation rapide de la concentration cytosolique d'une protéine lorsque l'expression des gènes qui cocodifie qu'il augmente.
Bien qu'il ne s'agisse pas d'un processus extrêmement courant dans ces organismes, la principale RNM produite lors de la transcription peut souffrir de maturation post-consistation par "coupe et épissage". Cependant, le plus courant est d'observer les ribosomes attachés au primaire transcrit qui le traduit en même temps qu'il est transcrit à partir de la séquence d'ADN correspondante.
Compte tenu de ce qui précède, la traduction dans de nombreux procaryotes commence par la fin 5 ', car l'extrémité 3' de l'ARNm reste liée à l'ADN de la moisissure (et se produit concomitante avec la transcription).
Régions non traduites
Les cellules procaryotes produisent également des RNM avec des régions non traduites qui sont connues sous le nom de "Shine-Dalgarno Box" et dont la séquence consensus est Aggagg. Comme il est évident, les régions UTR des bactéries sont considérablement plus courtes que celles des cellules eucaryotes, bien qu'elles exercent des fonctions similaires pendant la traduction.
Processus
Dans les bactéries et autres organismes procaryotes, le processus de traduction est assez similaire à celui des cellules eucaryotes. Il se compose également de trois phases: initiation, allongement et résiliation, qui dépendent de facteurs procaryotes spécifiques, différents de ceux utilisés par les eucaryotes.
L'allongement, par exemple, dépend des facteurs d'allongement appelés EF-TU et EF-TS, au lieu du facteur eucaryote g.
Les références
- Alberts, B., Johnson, un., Lewis, J., Raff, m., Roberts, k., & Walter, P. (2007). Biologie de la cellule moléculaire. Garland Science. New York, 1392.
- Argile, s. & Brown, W. (2008) Traduction: ADN en ARNm en protéine. Education de la nature 1 (1): 101.
- Griffiths, un. J., Wessler, s. R., Lewontin, R. C., Gelbart, w. M., Suzuki, D. T., & Miller, J. H. (2005). Une introduction à l'analyse génétique. Macmillan.
- Lodish, H., Berk, un., Kaiser, C. POUR., Krieger, m., Scott, M. P., Bretscher, un.,… & Matsudaira, P. (2008). Biologie des cellules moléculaires. Macmillan.
- Nelson, D. L., Lehninger, un. L., & Cox, M. M. (2008). Principes de lehninger de la biochimie. Macmillan.
- Rosenberg, L. ET., & Rosenberg, D. D. (2012). Gènes et génomes humains: science. Santé, société, 317-338.
- « Caractéristiques de Sorbus Aria, habitat, propriétés, culture
- Boletus aereus Caractéristiques, habitat, identification, recettes »

