Méthodes d'étude haplootype, diagnostics, maladies
- 4330
- 622
- Paul Dumas
UN Haplootype C'est une région du génome qui a tendance à hériter ensemble à travers plusieurs générations; En règle générale, tout est situé dans le même chromosome. Les haplootypes sont le produit d'une liaison génétique et restent intacts pendant la recombinaison génétique.
Le mot "haplotipo" dérive d'une combinaison du mot "haploïde" et du mot "génotype". "Haploïd" fait référence aux cellules avec un seul ensemble de chromosomes et "génotype" parle de la composition génétique d'un organisme.
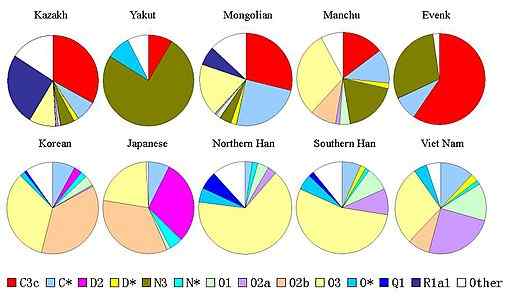
 Schéma de distribution des haplotypes chromosomes et des populations asiatiques (source: Moogalord [domaine public] via Wikimedia Commons) Après la définition, un haplotype peut décrire quelques gènes ou plus peut décrire un chromosome qui est complètement hérité d'un parent, comme c'est le cas du chromosome et des hommes.
Schéma de distribution des haplotypes chromosomes et des populations asiatiques (source: Moogalord [domaine public] via Wikimedia Commons) Après la définition, un haplotype peut décrire quelques gènes ou plus peut décrire un chromosome qui est complètement hérité d'un parent, comme c'est le cas du chromosome et des hommes.
Par exemple, lorsque les haplotypes partagent des gènes pour deux caractères phénotypiques différents, tels que la couleur des cheveux et la couleur des yeux, les individus qui ont un gène de couleur cheveux posséderont également l'autre gène pour la couleur des yeux.
Les haplootypes sont l'un des outils les plus utilisés aujourd'hui pour l'étude de la généalogie, pour retracer l'origine des maladies, pour caractériser la variabilité génétique et la phylogéographie des populations de différents types d'êtres vivants.
Il existe plusieurs outils pour l'étude des halootypes, l'un des plus utilisés aujourd'hui est "Carte haplotype«(HAPMAP), qui est une page Web qui permet de déterminer quels sont les segments du génome qui sont des haplotypes.
[TOC]
Méthodes d'étude
Les haplootypes représentent une occasion de comprendre l'héritage des gènes et leur polymorphisme. Avec la découverte de la technique "réaction en chaîne en polymérase" (PCR, anglais "Réaction en chaîne par polymérase») Il a largement avancé dans l'étude des haplotypes.
Il existe actuellement de nombreuses méthodologies pour l'étude des haplotypes, certains des plus forts sont:
Séquençage de l'ADN et détection de polymorphisme mononocléotidique (SNP)
Le développement des technologies de séquençage de nouvelle génération a représenté un grand saut pour l'étude des haplotypes. Les nouvelles technologies permettent aux variations de jusqu'à une seule base de nucléotides dans des régions spécifiques d'un haplotype.
Peut vous servir: ADN recombinant: technique, applications et fondationsEn bioinformatique, le terme haplotype est également utilisé pour désigner l'héritage d'un seul nucléotide polymorphismes (SNP) dans les séquences d'ADN.
La combinaison de programmes de bioinformatique avec la détection des haplotypes à l'aide de séquençage de nouvelle génération peut être identifié exactement la position, le remplacement et l'effet du changement de chaque base sur le génome d'une population.
Microsatellites (SSR)
Les microsatellites ou les SSR dérivent leur nom de l'anglais «Smettre en œuvre la répétition et Répétition en tandem courte". Ce sont des séquences nucléotidiques courtes qui sont répétées successivement dans une région du génome.
Il est courant de trouver des microsatellites à l'intérieur des haplotypes non codants, par conséquent, par la détection de variations du nombre de répétitions des microsatellites, différents allèles peuvent être observés dans les haplotypes des individus.
Des marqueurs moléculaires microsatellites ont été développés pour la détection d'haplotypes sans fin, à partir du sexate de plantes telles que la papaye (Carica papaya) jusqu'à la détection de maladies humaines telles que l'anémie falciforme.
Polymorphismes des fragments amplifiés (AFLP)
Cette technique combine l'amplification avec des réactions de PCR avec la digestion de l'ADN avec deux enzymes de restriction différentes. La technique détecte les loci polymorphes en haplootypes selon les différents sites de coupe de la séquence d'ADN.
Pour mieux illustrer la technique, imaginons trois fragments de tissu de la même longueur, mais coupés en différents endroits (ces fragments représentent trois fragments d'haplotypes amplifiés en utilisant la technique de PCR).
Au moment où le tissu est coupé, de nombreux morceaux de taille différente seront obtenus, car chaque tissu est coupé en différents endroits. Lors de la commande des fragments en fonction du type de tissu dont ils proviennent, nous pouvons observer où se trouvent les différences entre les tissus ou les haplotypes.
Diagnostic et maladies
Un avantage important de l'étude génétique des haplotypes est que ceux-ci restent presque intacts ou inchangés pendant des milliers de générations, ce qui permet d'identifier les ancêtres éloignés et chacune des mutations avec lesquelles les individus contribuent au développement de maladies.
Peut vous servir: variations phénotypiquesLes haplootypes dans l'humanité varient en fonction des races et, à partir de ce scoop, les gènes ont été détectés dans des haplotypes qui provoquent des maladies graves dans chacune des races humaines.
Dans le projet Hapmap Quatre groupes raciaux sont inclus: européen, yoruba nigérian, han chinois et japonais.
De cette façon, le projet Hapmap Il peut couvrir les différents groupes de populations et suivre l'origine et l'évolution de nombreuses maladies héréditaires qui affectent chacune des quatre races.
L'une des maladies qui est le plus fréquemment diagnostiquée à l'aide de l'analyse des haplotypes est l'anémie falciforme chez l'homme. Cette maladie est diagnostiquée en suivant la fréquence des haplotypes africains dans une population.
Être une maladie originaire d'Afrique, l'identification des haplotypes africains dans les populations vous permet de retracer facilement les personnes qui possèdent la mutation dans la séquence génétique pour la bêta globine des érythrocytes sous la forme d'un HOZ (caractéristique de la pathologie).
Exemples
Avec des haplotypes arbres phylogénétiques qui représentent des relations évolutives entre chacun des haplotypes trouvés dans un échantillon de molécules d'ADN homologues ou de la même espèce, dans une région qui a peu ou pas de recombinaison sont construites.
L'une des branches les plus étudiées à travers des haplotypes est l'évolution du système immunitaire humain. Les haplootypes qui code pour le récepteur de type péage (un composant clé du système immunitaire inné) ont été identifiés pour le génome de Néandertal et de Denisovanos.
Cela leur permet de tracer comment les séquences génétiques ont changé dans les populations des humains "modernes" en ce qui concerne les séquences d'haplotypes qui correspondent à des humains "ancestraux".
Construire un réseau de relations génétiques à partir d'un haplotype mitochondrial.
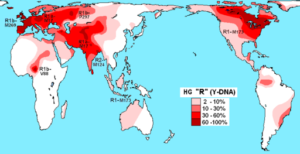 Distribution haplogroupe R (ADN-Y) dans les populations indigènes (Source: Maucioni [CC par 3.0 (https: // CreativeCommons.Org / licences / par / 3.0)], via Wikimedia Commons) La diversité des haplotypes est utilisée pour suivre et étudier la diversité génétique des animaux élevés en captivité. Ces techniques sont utilisées spécialement pour les espèces difficiles pour surveiller la faune.
Distribution haplogroupe R (ADN-Y) dans les populations indigènes (Source: Maucioni [CC par 3.0 (https: // CreativeCommons.Org / licences / par / 3.0)], via Wikimedia Commons) La diversité des haplotypes est utilisée pour suivre et étudier la diversité génétique des animaux élevés en captivité. Ces techniques sont utilisées spécialement pour les espèces difficiles pour surveiller la faune.
Les espèces animales telles que les requins, les oiseaux et les grands mammifères tels que les jaguars, les éléphants, entre autres, sont constamment évalués génétiquement à travers des haplotypes mitochondriaux pour surveiller l'état génétique des populations en captivité.
Les références
- Bahlo, m., Stankovich, J., Vitesse, t. P., Rubio, J. P., Burfoot, R. K., & Pied, s. J. (2006). Détetter le partage d'haplotypes larges du génome à l'aide de données Haplotype SNP ou microsatellites. Génétique humaine, 119 (1-2), 38-50.
- Dannemann, M., Andrés, un. M., & Kelso, J. (2016). Les introgresses de Néanderthal-eto, des haplotypes de type Denisovan, contribuent à la variation adaptative des récepteurs humains en forme de péage. The American Journal of Human Genetics, 98 (1), 22-33.
- De Vries, h. g., Van der meulen, m. POUR., Rozen, R., Halley, D. J., Scheffer, H., Leo, P.,… Et toi meerman, g. J. (mille neuf cent quatre vingt seize). Identité d'haplotype entre les individus qui partagent un allèle de mutation CFTR. Génétique humaine, 98 (3), 304-309
- Avaler, m. POUR., Leaver, un. L., Christiansen, F. T., Witt, c. S., Abraham, L. J., & Dawkins, R. L. (1992). Haplotypes ancestraux: Haplotypes du MHC de la population de conserve. Immunologie humaine, 34 (4), 242-252.
- Fellows, m. R., Hartman, t., Hermelin, D., Landau, G. M., Rosamond, F., & Rozenberg, L. (2009, juin). Inférence haplotype contrainte par des données haplotypes plausibles. Dans le symposium annuel sur la correspondance des motifs combinatoires (pp. 339-352). Springer, Berlin, Heidelberg.
- Gabriel, s. B., Schaffner, s. F., Nguyen, h., Moore, J. M., Roy, J., Blumenstiel, B.,... & liu-terroro, s. N. (2002). La structure des blocs d'haplotype dans le génome humain. Science, 296 (5576), 2225-2229.
- Consortium international HAPMAP. (2005). Une carte d'haplotypes du génome humain. Nature, 437 (7063), 1299.
- Wynne, R., & Wilding, C. (2018). Diversité des haplotypes d'ADN mithochondrial et origine des requins de tigres de sable captifs (Carcharias Taurus). Journal of Zoo and Aquarium Research, 6 (3), 74-78.
- Yoo, et. J., Tang, J., Kaslow, R. POUR., & Zhang, k. (2007). Inférence des haplotypes pour les données de génotype actuel à l'aide d'haplotypes et de modèles d'haplotypes identifiés par le. Bioinformatics, 23 (18), 2399-2406.
- Jeune, n. S. (2018). Anémie aplastique. The New England Journal of Medicine, 379 (17), 1643-1656.
- « Caractéristiques de l'héritage hollandique, fonctions des gènes, dégénérescence
- Caractéristiques plus importantes, habitat, propriétés, soins »

