Mécanismes de réplication de l'ADN, chez les procaryotes et les eucaryotes
- 807
- 34
- Anaïs Julien
La Réplication de ADN (Acide desoxyribonucléique) Il consiste à copier le génome, c'est-à-dire toutes les informations génétiques contenues dans l'ADN d'un organisme, pour produire deux copies identiques. Le génome a les informations nécessaires pour construire un organisme complet.
Avant la division cellulaire, la réplication de l'ADN se produit. Grâce à la méiose, des gamètes pour la reproduction sexuelle se produisent. Grâce à la mitose, le remplacement des cellules se produit (par exemple, la peau et le sang) et le développement (par exemple, les tissus et les organes).
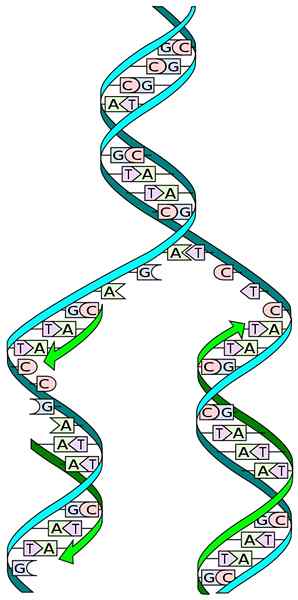
 Source: I, Madprime [CC0]
Source: I, Madprime [CC0] La connaissance de la structure d'ADN nous permet de comprendre la façon dont sa réplication se produit. La structure de l'ADN se compose d'une double hélice, composée de deux chaînes antipaallales de nucléotides successifs, dont les bases d'azote sont complétées spécifiquement.
Pendant la réplication, chacune des chaînes de la chaîne ADN double agit comme un moule pour la biosynthèse d'une nouvelle chaîne. Les deux chaînes nouvellement synthétisées ont des bases complémentaires des bases de la chaîne de moule: adénine (a) avec timina (t) et cytosine (c) avec de la guanine (g).
Plusieurs enzymes et protéines participent à la réplication de l'ADN. Par exemple, l'ouverture de l'hélice ADN double, le maintien de l'ADN ouvert et l'ajout de désoxyribonucléosidos-5'-triphosphate (DNTP) pour former la nouvelle chaîne.
[TOC]
La réplication de l'ADN est semi-conservatrice
Sur la base de la structure de l'ADN, Watson et Crick ont proposé que la réplication de l'ADN se produit de manière semi-conservatrice. Cela a été démontré par Meselson et Stahl en marquant l'ADN de Escherichia coli Avec l'isotope lourde de l'azote, quinzeN, suivant plusieurs générations le modèle de distribution dans un milieu de culture avec de l'azote léger, 14N.
Messelson et Stahl ont constaté que, dans la première génération, les deux molécules d'ADN de filles avaient, chaque molécule marquée d'une chaîne avec l'isotope d'azote lourd et un autre avec l'isotope léger. Contrairement à la molécule d'ADN parentale, qui avait les deux chaînes marquées de l'isotope lourd, quinzeN.
Dans la deuxième génération, 50% des molécules d'ADN étaient comme celles de la première génération, et les 50% restants n'avaient que de l'azote léger. L'interprétation de ce résultat est que l'hélice de double fille a une chaîne parentale (qui fonctionne comme un moule) et une nouvelle chaîne.
Le mécanisme de réplication semi-conservateur implique la séparation des chaînes d'ADN et l'accouplement des bases complémentaires au moyen de nucléotides successifs, produisant deux filles doubles filles filles.
Réplication dans les batteries
Début de la réplication de l'ADN dans les bactéries
L'ADN des bactéries se compose d'un chromosome circulaire et n'a qu'un seul site d'origine de la réplication. De ce site, la biosynthèse des deux filles se produit bidirectionnellement, formant deux fourches de réplication qui se déplacent dans des directions opposées à l'origine. En fin de compte, les fourches sont en terminant la réplication.
La réplication commence par l'union des protéines DNAA sur le site d'origine. Ces protéines forment à leur tour un complexe. Ensuite, les protéines HU et IHF sont liées.
Ensuite, les protéines DNAC sont jointes, ce qui fait rejoindre les hélicases de l'ADN. Ceux-ci aident à détendre l'ADN et à briser les liaisons hydrogène, formées en paires de bases. Ainsi, les deux chaînes se séparent encore plus, formant deux chaînes simples.
Peut vous servir: qu'est-ce que la permutation chromosomique?La topoisomérase II, ou ADN Girasa, se déplace devant l'ADN Helicy. Les protéines de liaison à l'ADN à chaîne unique (SSB) gardent les chaînes d'ADN séparées. Ainsi, la biosynthèse de la chaîne fille peut commencer.
Biosynthèse des filles des chaînes d'ADN dans les bactéries
L'enzyme Prima est responsable de la synthèse de chaînes d'ARN courtes appelées amorces, qui ont 10 à 15 nucléotides de longueur. L'ADN polymérase commence à ajouter du triphosphate de 5'.
Parce que les chaînes d'ADN sont antiparallèles, une amorce est synthétisée dans la chaîne de guidage et de nombreuses amorces dans la chaîne retardée. Pour cette raison, la biosynthèse de la chaîne retardée est discontinue. Bien que les chaînes d'ADN soient antiparallèles, la fourche de réplication se déplace dans une direction.
L'ADN polymérase est responsable de la formation de liaisons covalentes entre les nucléotides adjacents des chaînes nouvellement synthétisées, dans une direction de 5'®3 '. Dans ET. coli, Il existe cinq ADN polymérase: l'ADN polymérases I et III effectuent une réplication de l'ADN; et les ADN polymérases II, IV et V sont responsables de la réplication et de la réplication de l'ADN endommagé.
La majeure partie de la réplication est faite par l'ADN polymérase III, qui est une holoenzyme qui a 10 sous-unités différentes avec plusieurs fonctions dans la réplication de l'ADN. Par exemple, la sous-unité alpha est responsable de la création de liaisons entre les nucléotides.
Un complexe enzymatique est responsable de la réplication de l'ADN dans les bactéries
L'hélicase de l'ADN et du prima est jointe pour former un complexe appelé primosoma. Cela se déplace le long de l'ADN, agissant de manière coordonnée pour séparer les deux chaînes parentales, synthétisant les amorces à chaque intervalle de la chaîne retardée.
Le primosome se lie physiquement à l'ADN polymérase III et forme le replisome. Deux ADN polymérases III sont responsables de la réplication de l'ADN du guide et des chaînes retardées. En ce qui concerne l'ADN polymérase III, la chaîne retardée forme une boucle, ce qui permet à l'ajout de nucléotides à cette chaîne de se produire dans la même direction de la chaîne de guidage.
L'ajout de nucléotides à la chaîne de guidage est continu. Dans le retard, il est discontinu. Des fragments de 150 nucléotides de longueur sont formés, appelés fragments d'Okazaki.
L'activité d'exonucléase 5 '-> 3' de l'ADN polymérase I est responsable de l'élimination des amorces et du remplissage, ajoutant des nucléotides. Une enzyme ligase scelle les lacunes entre les fragments. La réplication se termine lorsque les deux hoquillas de réplication sont dans une séquence d'achèvement.
La protéine que vous vous lie à la séquence de terminaison, arrêtant le mouvement de la fourche de réplication. La topoisomérase II permet la séparation des deux chromosomes.
Desoxiribonucléotides trypphosphates sont utilisés par l'ADN polymérase
Dexinucléoside Tryphosphate (DNTP) contient trois groupes de phosphate unis au carbone 5 'de Deoxyribosa. Les DNTP (DATP, DTTP, DGTP et DCTP) rejoignent la chaîne de moule suivant la règle AT / GC.
Peut vous servir: plésiomorphieL'ADN polymérase catalyse la réaction suivante: Le groupe hydroxyle (-OH) 3 'du nucléotide de la chaîne de croissance réagit avec l'alpha phosphate du DNTP entrant, libérant le pyrophosphate inorganique (PPI) (PPI). L'hydrolyse PPI produit de l'énergie pour la formation de la liaison covalente, ou liaison phosphodiéster, entre les nucléotides de la chaîne de croissance.
Mécanismes qui assurent la fidélité de la réplication de l'ADN
Pendant la réplication de l'ADN, l'ADN polymérase III fait une erreur pour 100 millions de nucléotides. Bien que la probabilité d'erreur soit très faible, il existe des mécanismes qui garantissent la fidélité dans la réplication de l'ADN. Ces mécanismes sont:
1) Stabilité dans l'apar de base. L'énergie de liaison hydrogène entre AT / GC est supérieure à celle des paires de bases erronées.
2) Structure du site actif de l'ADN polymérase. L'ADN polymérase catalyse préférentiellement les nucléotides avec des bases correctes dans la chaîne opposée. Un mauvais accouplement des bases provoque une distorsion de l'hélice ADN double, ce qui empêche le nucléotide incorrect d'occuper le site actif de l'enzyme.
3) Test de lecture. L'ADN polymérase identifie les nucléotides erronés incorporés et les élimine de la chaîne fille. L'activité d'exonucléase de l'ADN polymérase rompt les liaisons phosphodiéster entre les nucléotides à l'extrémité 3 'de la nouvelle chaîne.
Réplication de l'ADN chez les eucaryotes
Contrairement à la réplication dans les procaryotes, dont la réplication commence dans un seul site, la réplication eucaryote commence dans plusieurs sites d'origine et la fourche de réplication se déplace bidirectionnellement. Par la suite, toutes les fourchettes de réplication sont fusionnées, formant deux chromatides sœurs jointes au Centromère.
Les eucaryotas ont de nombreux types d'ADN polymérase, dont les noms utilisent des lettres grecques. ADN polymérase α former un complexe avec prima. Ce complexe synthétise des amorces courtes constituées de 10 nucléotides d'ARN suivis de 20 à 30 nucléotides d'ADN.
Puis ADN polymérase ε soit δ catalyse l'allongement de la chaîne fille de l'amorce. ADN polymérase ε Il est impliqué dans la synthèse de la chaîne principale, tandis que l'ADN polymérase δ Synthétiser la chaîne retardée.
ADN polymérase δ Allongez le fragment d'Okazaki gauche jusqu'à atteindre l'amorce d'ARN droit, produisant un court soulèvement de l'amorce ("rabat court"). Contrairement aux procaryotes, où un ADN de polymérase élimine l'amorce, dans les eucaryotes, une enzyme du volet endonucléase élimine l'amorce d'ARN.
Ensuite, un ADN ligase scelle les fragments d'ADN qui sont adjacents. L'achèvement de la réplication a lieu avec la dissociation des protéines de la fourche de réplication.
La Réplication de l'ADN dans les eucaryotes et le cycle cellulaire
La réplication chez les eucaryotes se produit dans la phase S du cycle cellulaire. Les molécules d'ADN répliquées sont séparées dans deux cellules filles pendant la mitose. Les phases G1 et G2 séparent la phase S et la mitose. La progression à travers chaque phase du cycle cellulaire est hautement régulée par les kinases, les phosphatases et les protéases.
Dans la phase G1 du cycle cellulaire, le complexe de reconnaissance d'origine (OCR) rejoint le site d'origine. Cela induit l'union des héliques MCM et d'autres protéines, telles que CDC6 et CDT1, pour former un complexe de pré-réplication (PRERC). Las Helicase MCM rejoint la chaîne de guides.
Peut vous servir: gènes liésDans la phase S, PRERC devient un site de réplication actif. Les protéines OCR, CDC6 et CDT1 sont libérées, et l'hélicase MCM se déplace en direction de 3 'à 5'. Une fois la réplication terminée, cela sera redémarré dans le cycle cellulaire suivant.
Réplication des extrémités des chromosomes chez les eucaryotes
Les extrémités des chromosomes sont appelées télomères, qui se composent de séquences répétées en tandem, et d'une région 3 'qui excelle, de 12 à 16 nucléotides de longueur.
L'ADN polymérase n'est pas en mesure de reproduire l'extrémité 3 'des chaînes d'ADN. En effet, l'ADN polymérase ne peut synthétiser l'ADN que dans l'adresse 5'-3 ', et ne peut allonger que des chaînes préexistantes, sans pouvoir synthétiser une amorce dans cette région. Par conséquent, les télomères sont raccourcis dans chaque tour de réplication.
L'enzyme de télomérase empêche le raccourcissement des télomères. La télomérase est une enzyme qui a des sous-unités de protéines et d'ARN (troisième). Ce dernier se lie aux séquences répétées de l'ADN et permet à la télomérase de se joindre à l'extrémité 3 'du télomère.
Une séquence d'ARN derrière l'union située comme moule pour la synthèse d'une séquence de six nucléotides (polymérisation) à la fin de la chaîne d'ADN. L'allongement du télomère est catalysé par des sous-unités de télomérase, appelée transcriptase de télomérase inverse (TERT).
Après la polymérisation, la translocalisation a lieu, composée du mouvement de la télomérase à une nouvelle extrémité de la chaîne d'ADN, rejoignant six autres nucléotides jusqu'à la fin.
Les fonctions d'autres ADN polymérases dans les eucaryotes
ADN polymérase β Il a un rôle important dans l'élimination des bases incorrectes de l'ADN, mais il n'est pas impliqué dans la réplication de l'ADN.
De nombreux ADN de polymérase découverts appartiennent au groupe polymérase «translésion-repliement». Ces polymérases sont responsables de la synthèse des chaînes complémentaires dans une région d'ADN endommagée.
Il existe plusieurs types de polymérases "translésion-reproduction". Par exemple, l'ADN polymérase η Vous pouvez vous reproduire sur Timina Dímeros, qui sont produites par la lumière UV.
Réplication de l'ADN dans les archéobactéries
La réplication de l'ADN archéobactérie est similaire à celle donnée dans les eucaryotes. Cela est dû à ce qui suit: 1) les protéines qui participent à la réplication ressemblent davantage à celles des eucaryotes qu'à celles des procaryotes; et 2) bien qu'il n'y ait qu'un seul site de réplication comme dans les procaryotes, sa séquence est similaire au site d'origine eucaryote.
La similitude de la réplication entre les arches et les eucaryotes soutient l'idée que les deux groupes sont phylogénétiquement plus liés les uns aux autres que n'importe lequel d'entre eux avec les procaryotes.
Les références
- Brooker, R. J. 2018. Analyse et principes de génétique. McGraw-Hill, New York.
- Hartwell, L. H., Goldberg, M. L., Fischer, J. POUR., Capot, L. 2018. Génétique - des gènes génomes. McGraw-Hill, New York.
- Kušić-tišma, J. 2011. Aspects fondamentaux de la réplication de l'ADN. Intech Open Access, Croatie.
- Lewis, R., 2015. Concepts et applications de génétique humaine. McGraw-Hill, New York.
- Pierce, b. POUR. 2005. Génétique - une approche conceptuelle. W. H. Freeman, New York.

